WST-1 Cell Proliferation Reagentto Avoid Off-Target Effects of RNAi
RNA interference(RNAi)의 발견
포유류 유전자 발현을 억제할수 있게 되었고 유전자 발현을 억제하여 유전자 기능을 연구하는 방법으로 연구자들에게 선호 되었다. 또한 RNAi 기술은 다양한 치료제 개발에서 엄청난 가능성을 보여줬으나 RNAi 염기서열이 특정 타겟을 억제하지 않고 다른 유전자도 억제한 경우(off-target effect)도 종종 보고되었다.
RNAi의 off-target을 방지하는 것은 세포내 독성을 방지하고 RNAi 실험 결과를 정확하게 해석하기 위해 매우 중요하다. RNAi 제작시 off-target 발생을 최소화 하기 위해 BLAST와 Smith-Waterman 분석과 같은 다양한 알고리즘이 도입되었지만, 실험 결과를 통해 bioinformatics방법은 off-target 방지책이 아니라는 것이 확인되었다. 반면 3’의 UTR에 있는 short seed sequences의 확인으로 offtarget 될 가능성을 실제보다 높게 평가했다는 주장이 있다. 직접적인 RNAi 특이성 정보를 얻기 위해 genome-wide expression 변화를 확 인하는 microarray 분석을 진행하였다. 그러나 이 분석법은 복잡하 고 많은 시간이 소요되며, 많은 후보 RNAi를 스크리닝 하기에 가격적인 부담이 있다.
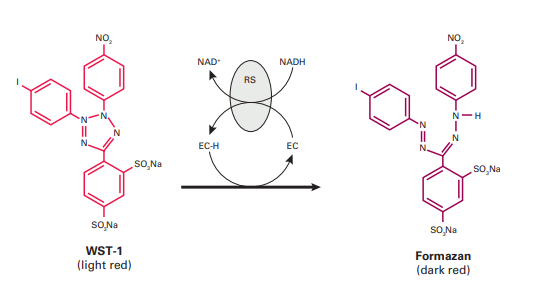
Off -target 현상은 세포 증식 억제나 독성과 같은 세포 활성의 변화가 나타나기 때문에, 세포 활성 모니터링을 통해 RNAi 영향력을 Premixed WST-1 Cell Proliferation Reagent로 확인할 수 있다. 이러한 비색 분석은 tetrazolium salt WST-1(밝은 빨강색)이 분해되 어 formazan(어두운 빨강색)이 되는 특징을 이용한 것이다
이 반응은 살아있는 세포의 미토콘드리아 환원효소에 의해 일어나며, multiplate reader를 이용하여 흡광도 420-480nm(Amax=450 nm)로 측정할 수 있다. Formazan 생산량은 대사작용이 활성화되어 있는 세포 와 비례한다
간염(Hepatitis C) 치료제 개발을 위한 후보
RNAi 서열 확인 대다수의 RNA 바이러스가 복제과정에서 돌연변이가 잘 발생하기 때문에, 서열 특이적인 RNAi를 기반으로 한 항바이러스제의 개발이 어렵다. 수백개의 viral isolate의 보존서열로부터 모든 HCV 유전자형 에서 특이적으로 적용되는 30개의 RNAi 후보를 선발하였다. 각 후보 RNAi의 발현 억제 기능을 확인하기 위해 siRNA duplexer를 합성하 였다. 최근까지 조직을 이용한 HCV 증식이 어려웠기 때문에 siRNA는 HCV replicon 모델이나 reporter assay와 같은 다른 시스템을 이용하 여 연구되었다. Reporter assay는 세포에 luciferase reporter plasmid 를 co-trasfection하여 siRNA 활성을 확인하는 방법으로, HCV의 약 100bp 서열이 luciferase open reading frame의 하위부분과 poly A tail 상류부분에 삽입된다. 이후, 삽입된 서열에서 luciferase reporter mRNA가 절단되므로 luminescence가 감소된다. 감소한 luminescence를 측정하여 RNAi 저해 효과를 확인할 수 있다.
Luciferase-based reporter와 replicon system assay를 이용하였을 때, 30개 siRNA 중 20개 siRNA 가 활성을 억제하는 것을 확인하였다. 그 후, 후보 siRNA는 기능항진 으로 알려진 농도에 따른 RNAi 억제 능력을 바탕으로 순위를 정했다. 후보 유전자를 줄이기 위해, Premixed WST-1 Cell Pro-liferation Reagent를 이용하여 저렴하고 빠르게 off-target된 siRNA를 제거하 였다
특히 세포내 타겟 유전자를 억제시키면 세포 독성과 활성 억제를 나타내 는 siRNA를 중점으로 진행하였다. 실제로 세포 분화에 필수적인 유전자 확인을 위해 진행한 RNAi based screening에서(Kittler et al.) WST1 assay를 이용하였다. 이 연구는 15,497 인간 유전자 cDNA library를 이용하여 제작된 endoribonuclease-prepared short interfering(esi) RNA genome-scale library를 스크리닝하여, 세포 활성을 억제 시키는 RNAi를 확인하였다(5). 실제 세포 내에는 바이러스 염기서열이 존재하지 않기 때문에, 감염되지 않은 세포에서 바이러스 특이적 siRNA로 인한 세포활성 감소는 세포 내 유전자의 억제로 인한 결과와 같다고 판단하였 다. WST-1 assay의 감도와 측정범위를 테스트하기 위해 감염시키지 않은 Huh-7 세포를 다양한 농도로 준비한 후, 72 시간 동안 세포 배양하여 WST-1 assay를 진행하였다. 그 결과, 농도 차이에 따라 세포 활성 정도 가 확연하게 나타나 간편하게 구별 가능했다